分子類似性検索 (Molecular Similarity Search)
概要
分子類似性検索は、既知の活性化合物と構造的に類似した新しい化合物を発見するための重要な創薬手法です。本ノートブックでは、様々な類似性指標とフィンガープリント手法を用いて、効率的な分子類似性検索システムを構築します。
学習目標
- 分子類似性の概念と創薬における重要性を理解する
- 各種フィンガープリント手法の特徴と使い分けを学ぶ
- Tanimoto係数をはじめとする類似性指標を習得する
- バーチャルスクリーニングの実装と評価方法を学ぶ
- 化学空間の可視化と分析手法を理解する
補助資料
初心者の方へ: この内容が難しく感じる場合は、以下の補助資料をご参照ください:
- 分子類似性検索 初心者ガイド: 基本概念を日常的な例えで解説
- 分子類似性検索 用語集: 専門用語の分かりやすい説明と参考リンク
これらの資料は、文系出身の方や化学・情報学の予備知識が少ない方でも理解できるよう工夫されています。
ダウンロード
|
|
ライブラリのインポートが完了しました
1. 分子フィンガープリントの種類と比較
分子フィンガープリントは分子の構造情報をビットベクトルで表現する手法です。用途に応じて適切なフィンガープリントを選択することが重要です。
|
|
フィンガープリント生成テスト:
==================================================
アスピリン:
Morgan_r2: 24/1024 bits on
Morgan_r3: 31/1024 bits on
RDKit: 319/1024 bits on
MACCS: 21/167 bits on
AtomPair: 68/1024 bits on
TopologicalTorsion: 17/1024 bits on
イブプロフェン:
Morgan_r2: 25/1024 bits on
Morgan_r3: 30/1024 bits on
RDKit: 214/1024 bits on
MACCS: 15/167 bits on
AtomPair: 91/1024 bits on
TopologicalTorsion: 21/1024 bits on
ナプロキセン:
Morgan_r2: 34/1024 bits on
Morgan_r3: 45/1024 bits on
RDKit: 337/1024 bits on
MACCS: 22/167 bits on
AtomPair: 108/1024 bits on
TopologicalTorsion: 28/1024 bits on
インドメタシン:
Morgan_r2: 39/1024 bits on
Morgan_r3: 54/1024 bits on
RDKit: 775/1024 bits on
MACCS: 41/167 bits on
AtomPair: 175/1024 bits on
TopologicalTorsion: 41/1024 bits on
ジクロフェナク:
Morgan_r2: 29/1024 bits on
Morgan_r3: 41/1024 bits on
RDKit: 392/1024 bits on
MACCS: 28/167 bits on
AtomPair: 132/1024 bits on
TopologicalTorsion: 29/1024 bits on
セレコキシブ:
Morgan_r2: 39/1024 bits on
Morgan_r3: 52/1024 bits on
RDKit: 724/1024 bits on
MACCS: 49/167 bits on
AtomPair: 250/1024 bits on
TopologicalTorsion: 46/1024 bits on
[17:00:43] DEPRECATION WARNING: please use MorganGenerator
[17:00:43] DEPRECATION WARNING: please use MorganGenerator
[17:00:43] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:43] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:43] DEPRECATION WARNING: please use MorganGenerator
[17:00:43] DEPRECATION WARNING: please use MorganGenerator
[17:00:43] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:43] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:43] DEPRECATION WARNING: please use MorganGenerator
[17:00:43] DEPRECATION WARNING: please use MorganGenerator
[17:00:43] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:43] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:43] DEPRECATION WARNING: please use MorganGenerator
[17:00:43] DEPRECATION WARNING: please use MorganGenerator
[17:00:43] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:43] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:43] DEPRECATION WARNING: please use MorganGenerator
[17:00:43] DEPRECATION WARNING: please use MorganGenerator
[17:00:43] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:43] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:43] DEPRECATION WARNING: please use MorganGenerator
[17:00:43] DEPRECATION WARNING: please use MorganGenerator
[17:00:43] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:43] DEPRECATION WARNING: please use TopologicalTorsionGenerator
2. 類似性指標の比較
分子類似性を定量化するための様々な指標があります。最も一般的なTanimoto係数に加えて、Dice係数、Tversky係数などを比較します。
|
|
基準分子: アスピリン
============================================================
イブプロフェン:
Tanimoto : 0.195
Dice : 0.327
Tversky_0.8_0.2: 0.331
Tversky_0.5_0.5: 0.327
ナプロキセン:
Tanimoto : 0.261
Dice : 0.414
Tversky_0.8_0.2: 0.462
Tversky_0.5_0.5: 0.414
インドメタシン:
Tanimoto : 0.189
Dice : 0.317
Tversky_0.8_0.2: 0.370
Tversky_0.5_0.5: 0.317
ジクロフェナク:
Tanimoto : 0.205
Dice : 0.340
Tversky_0.8_0.2: 0.360
Tversky_0.5_0.5: 0.340
セレコキシブ:
Tanimoto : 0.105
Dice : 0.190
Tversky_0.8_0.2: 0.222
Tversky_0.5_0.5: 0.190
3. フィンガープリント手法の性能比較
|
|
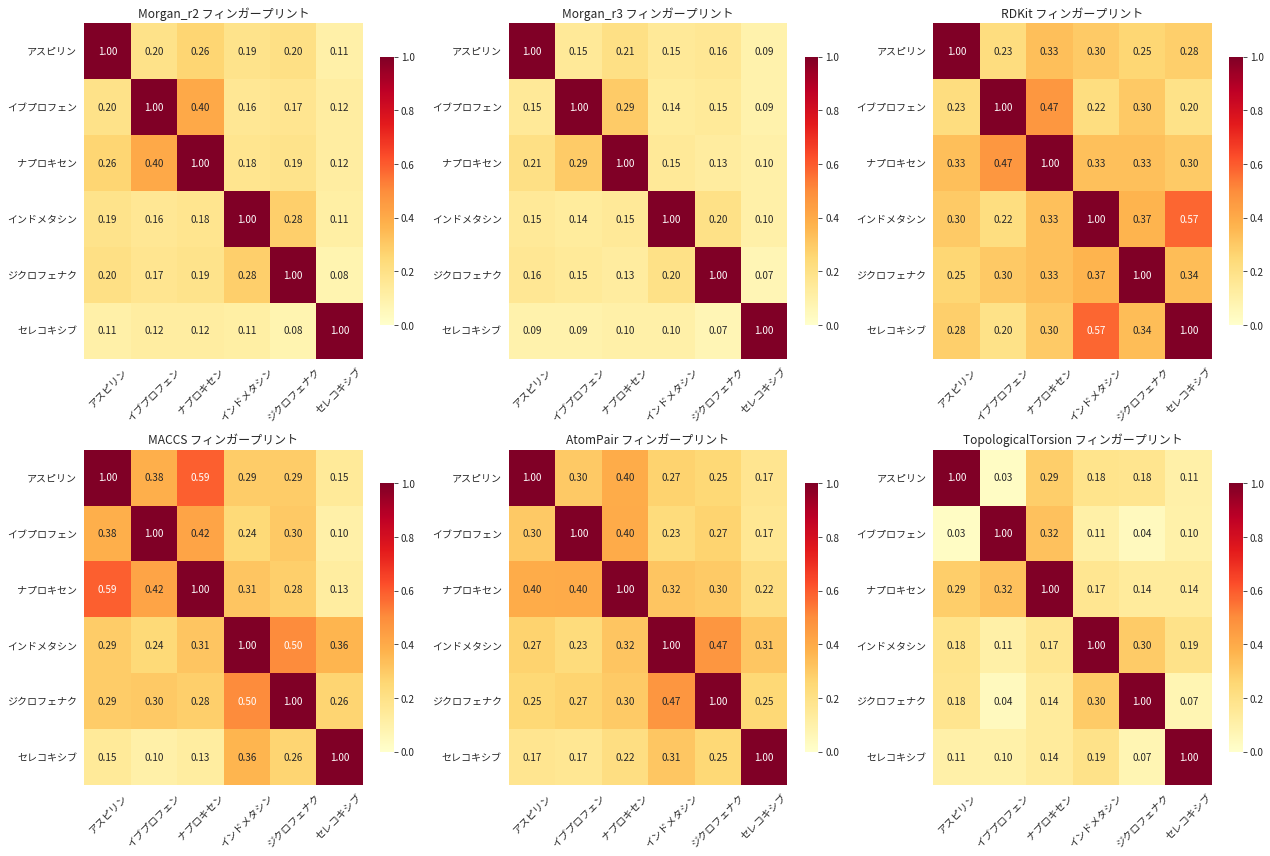
フィンガープリント手法の特徴分析:
==================================================
Morgan_r2 : 平均類似度 0.186 ± 0.080
Morgan_r3 : 平均類似度 0.146 ± 0.054
RDKit : 平均類似度 0.322 ± 0.093
MACCS : 平均類似度 0.308 ± 0.129
AtomPair : 平均類似度 0.289 ± 0.081
TopologicalTorsion : 平均類似度 0.157 ± 0.087
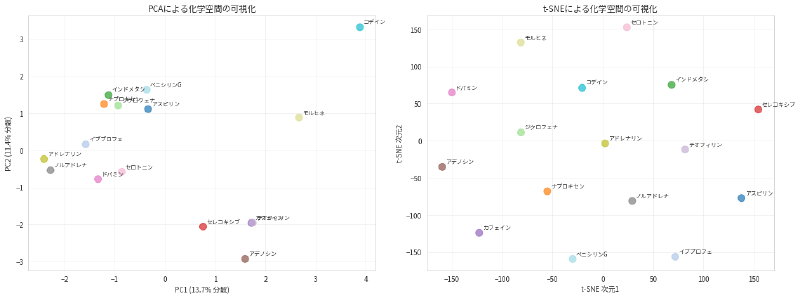
4. 化学空間の可視化
分子フィンガープリントを用いて化学空間を可視化し、分子間の関係性を理解します。t-SNEとPCAを用いて次元削減を行います。
|
|
分析対象分子数: 16
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use MorganGenerator
[17:00:46] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:46] DEPRECATION WARNING: please use TopologicalTorsionGenerator
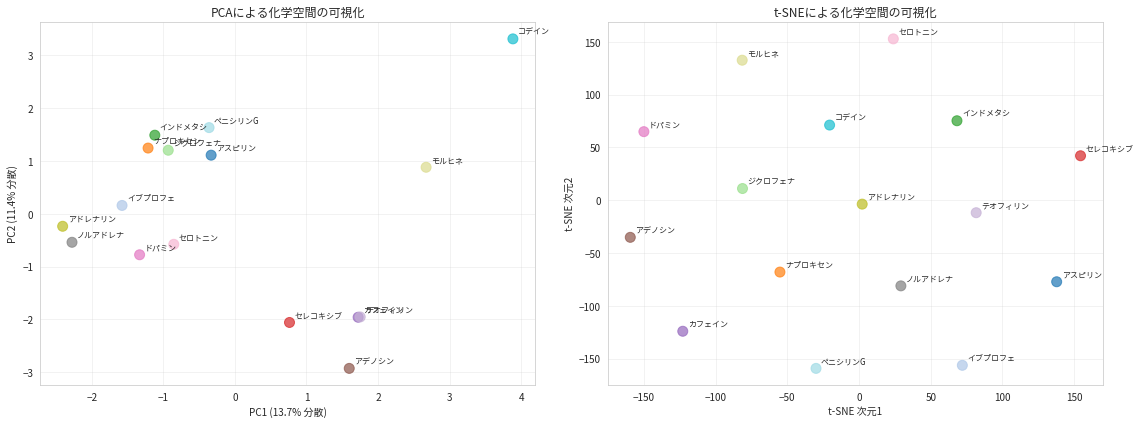
PCA: 第1・第2主成分で 25.1% の分散を説明
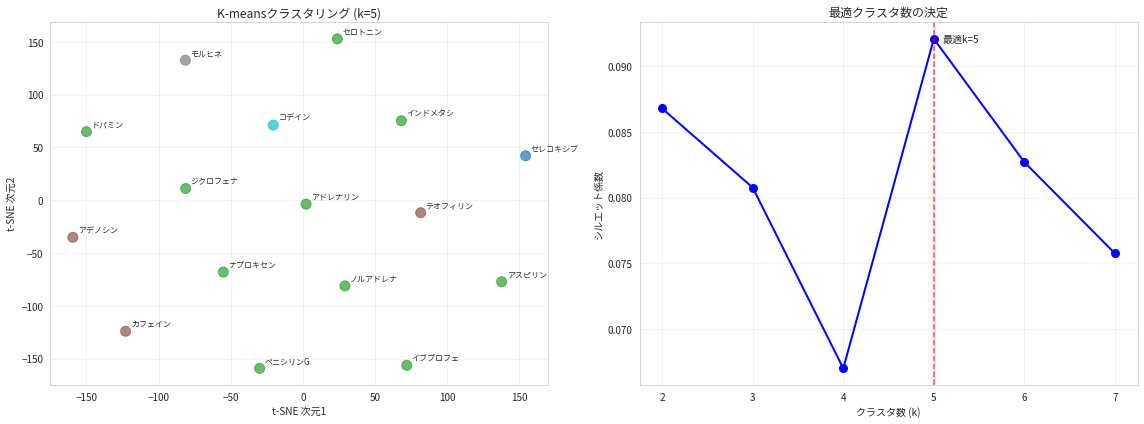
5. クラスタリング分析
分子の構造的類似性に基づいてクラスタリングを行い、化学的に関連する分子群を特定します。
|
|
Butinaクラスタリング結果:
========================================
クラスタ 1: 2 分子
- ペニシリンG, セレコキシブ
クラスタ 2: 1 分子
- コデイン
クラスタ 3: 1 分子
- モルヒネ
クラスタ 4: 1 分子
- アドレナリン
クラスタ 5: 1 分子
- ノルアドレナリン
クラスタ 6: 1 分子
- セロトニン
クラスタ 7: 1 分子
- ドパミン
クラスタ 8: 1 分子
- アデノシン
クラスタ 9: 1 分子
- テオフィリン
クラスタ 10: 1 分子
- カフェイン
クラスタ 11: 1 分子
- ジクロフェナク
クラスタ 12: 1 分子
- インドメタシン
クラスタ 13: 1 分子
- ナプロキセン
クラスタ 14: 1 分子
- イブプロフェン
クラスタ 15: 1 分子
- アスピリン
最適なクラスタ数 (K-means): 5 (シルエット係数: 0.092)
6. バーチャルスクリーニングの実装
実際の創薬研究で用いられるバーチャルスクリーニングを実装します。既知の活性化合物に類似した化合物を大規模ライブラリから検索します。
|
|
バーチャルスクリーニング結果:
============================================================
基準分子数: 3
スクリーニング化合物数: 8
有効結果数: 8
スクリーニング結果(類似度順):
SMILES 最大類似度 平均類似度 ヒット判定
4 CC(CC1=CC=CC=C1)C(=O)O 0.516 0.376 True
5 COC1=CC=C(C=C1)C(=O)C2=CC=CC=C2 0.343 0.279 False
0 O=C(O)CC1=CC=CC=C1NC2=C(Cl)C=CC=C2Cl 0.205 0.189 False
1 CC1=C(C2=CC=CC=C2N1C(=O)C3=CC=C(C=C3)Cl)CC(=O)O 0.189 0.177 False
3 C1=CC(=C(C=C1CCN)O)O 0.179 0.133 False
7 NC1=CC=C(C=C1)S(=O)(=O)NC2=CC=CC=C2 0.167 0.130 False
6 CC1=CC=C(C=C1)S(=O)(=O)NC2=NC(=CS2)C 0.121 0.114 False
2 CN1C=NC2=C1C(=O)N(C(=O)N2C)C 0.114 0.100 False
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:47] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:47] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:47] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:47] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:47] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:47] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:47] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:47] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:47] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:47] DEPRECATION WARNING: please use TopologicalTorsionGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use AtomPairGenerator
[17:00:47] DEPRECATION WARNING: please use TopologicalTorsionGenerator
7. スクリーニング結果の可視化と分析
|
|
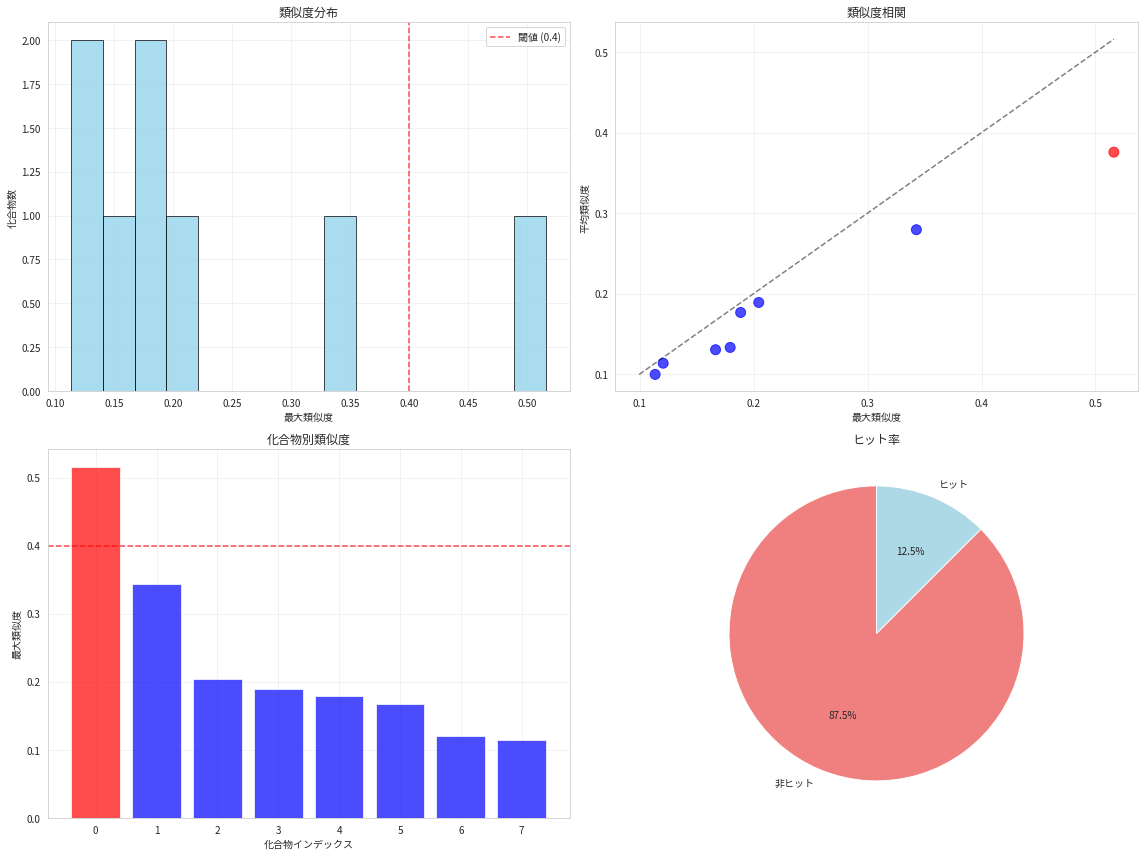
スクリーニング統計:
==============================
総化合物数: 8
ヒット数: 1
ヒット率: 12.5%
最大類似度 - 平均: 0.229 ± 0.136
平均類似度 - 平均: 0.187 ± 0.095
8. 多様性を考慮した化合物選択
創薬研究では類似性だけでなく、化合物の多様性も重要です。MaxMinアルゴリズムを用いて多様性を考慮した化合物選択を行います。
|
|
多様性を考慮した化合物選択:
========================================
ライブラリサイズ: 15
選択された 5 化合物:
1. 化合物_1: CC(=O)OC1=CC=CC=C1C(=O)O
2. 化合物_10: C1=CC2=C(C=C1O)C(=CN2)CCN
3. 化合物_8: CN1C=NC2=C1C(=O)N(C(=O)N2C)C
4. 化合物_6: CC1=CC=C(C=C1)S(=O)(=O)NC2=NC(=CS2)C
5. 化合物_5: CC1=C(C2=CC=CC=C2N1C(=O)C3=CC=C(C=C3)Cl)CC(=O)O
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
[17:00:47] DEPRECATION WARNING: please use MorganGenerator
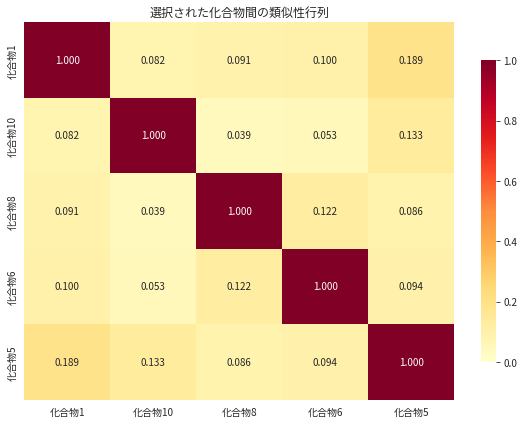
多様性統計:
化合物間平均類似度: 0.099 ± 0.040
最小類似度: 0.039
最大類似度: 0.189
まとめ
本ノートブックでは、分子類似性検索の様々な手法について学習しました:
主要なポイント
-
フィンガープリント手法:
- Morgan (ECFP)、RDKit、MACCS、AtomPair、TopologicalTorsion
- 用途に応じた適切な手法の選択が重要
-
類似性指標:
- Tanimoto係数:最も一般的で対称的
- Dice係数:Tanimoto係数より高い値を示す傾向
- Tversky係数:非対称的な類似性評価が可能
-
化学空間の可視化:
- PCA:線形次元削減、解釈しやすい
- t-SNE:非線形次元削減、クラスタ構造を保持
-
クラスタリング:
- Butinaクラスタリング:化学的に意味のあるクラスタ
- K-meansクラスタリング:効率的だが球状クラスタを仮定
-
バーチャルスクリーニング:
- 既知活性化合物からの類似化合物探索
- 閾値設定とヒット率の最適化
-
多様性選択:
- MaxMinアルゴリズム:多様性を考慮した化合物選択
- 類似性と多様性のバランス
創薬への応用
- リード化合物最適化: 構造活性相関の理解
- 化合物ライブラリ設計: 多様性を考慮した合成標的選択
- 薬物再配置: 既存薬物の新たな適応症探索
- 副作用予測: 類似化合物の既知副作用からの推定
次のステップ
- 活性データを用いた教師ありモデル構築
- 3D分子構造を考慮した類似性評価
- 機械学習による類似性指標の学習
- 大規模データベースでの高速検索アルゴリズム
|
|
分子類似性検索学習の統計情報:
==================================================
解析フィンガープリント種類: 6
解析分子数: 16
バーチャルスクリーニング対象: 8
多様性選択ライブラリ: 15
最終選択化合物数: 5
各手法の特徴:
------------------------------
• Morgan フィンガープリント: 環境情報を考慮、半径可変
• MACCS キー: 166の構造パターン、高速
• RDKit フィンガープリント: パス情報、構造多様性
• Tanimoto係数: 対称的、一般的
• バーチャルスクリーニング: 効率的な候補化合物探索
• 多様性選択: MaxMinアルゴリズム、探索空間最大化
学習が完了しました!