記述子による薬らしさの表現 (Drug-likeness Descriptors)
概要
薬らしさ(Drug-likeness)とは、ある化合物が薬物として適した性質を持つ度合いを表す概念です。本ノートブックでは、分子記述子を用いて薬らしさを定量的に評価する手法について学習し、実際の薬物データベースを用いた分析を行います。
学習目標
- 薬らしさの概念と創薬における重要性を理解する
- Lipinski’s Rule of Fiveとその拡張ルールを習得する
- QED(Quantitative Estimate of Drug-likeness)の計算と解釈を学ぶ
- ADMETパラメータによる薬物動態予測を理解する
- 薬物と非薬物の判別手法を実装する
補助資料
初心者の方へ: この内容が難しく感じる場合は、以下の補助資料をご参照ください:
- 記述子による薬らしさ 初心者ガイド: 基本概念を日常的な例えで解説
- 記述子による薬らしさ 用語集: 専門用語の分かりやすい説明と参考リンク
これらの資料は、文系出身の方や化学・情報学の予備知識が少ない方でも理解できるよう工夫されています。
ダウンロード
|
|
ライブラリのインポートが完了しました
<frozen importlib._bootstrap>:228: RuntimeWarning: to-Python converter for boost::shared_ptr<RDKit::FilterHierarchyMatcher> already registered; second conversion method ignored.
1. 基本的な薬物記述子の計算
まず、薬らしさの評価に重要な基本的な分子記述子を計算します。これらの記述子は、分子の物理化学的性質を数値化し、薬物として適した特性を評価するために使用されます。
|
|
薬物記述子の計算結果:
================================================================================
化合物名 SMILES 分子量 \
0 アスピリン CC(=O)OC1=CC=CC=C1C(=O)O 180.16
1 イブプロフェン CC(C)CC1=CC=C(C=C1)C(C)C(=O)O 206.28
2 パラセタモール CC(=O)NC1=CC=C(C=C1)O 151.16
3 ペニシリンG CC1(C(N2C(S1)C(C2=O)NC(=O)CC3=CC=CC=C3)C(=O)O)C 334.40
4 アモキシシリン CC1(C(N2C(S1)C(C2=O)NC(=O)C(C3=CC=C(C=C3)O)N)C... 365.41
5 プロプラノロール CC(C)NCC(COC1=CC=CC2=C1C=CC=N2)O 260.34
6 アテノロール CC(C)NCC(COC1=CC=C(C=C1)CC(=O)N)O 266.34
7 モルヒネ CN1CCC23C4C1CC5=C2C(=C(C=C5)O)OC3C(C=C4)O 285.34
8 カフェイン CN1C=NC2=C1C(=O)N(C(=O)N2C)C 194.19
9 ジアゼパム CN1C(=O)CN=C(C2=C1C=CC(=C2)Cl)C3=CC=CC=C3 284.75
10 ドキソルビシン COC1=C(C=C2C(=C1)C(=O)C3=C(C2=O)C(=CC=C3O)O)O 286.24
11 タモキシフェン CCC(=C(C1=CC=CC=C1)C2=CC=C(C=C2)OCCN(C)C)C3=CC... 371.52
12 メトホルミン CN(C)C(=N)NC(=N)N 129.17
13 フルオキセチン CNCCC(C1=CC=CC=C1)OC2=CC=C(C=C2)C(F)(F)F 309.33
14 セルトラリン CNC1CCC(C2=CC=CC=C12)C3=CC(=C(C=C3)Cl)Cl 306.24
15 カフェイン酸 C1=CC(=C(C=C1C=CC(=O)O)O)O 180.16
16 ベンゼン C1=CC=CC=C1 78.11
17 トルエン CC1=CC=CC=C1 92.14
18 エタノール CCO 46.07
19 グルコース C(C1C(C(C(C(O1)O)O)O)O)O 180.16
20 アミノ酸_グリシン C(C(=O)O)N 75.07
21 脂肪酸_パルミチン酸 CCCCCCCCCCCCCCCC(=O)O 256.43
Heavy_Atoms LogP LogS HBD HBA TPSA PSA RotBonds RingCount \
0 13 1.31 -0.92 1 3 63.60 63.60 2 1
1 15 3.07 -2.15 1 1 37.30 37.30 4 1
2 11 1.35 -0.95 2 2 49.33 49.33 1 1
3 23 0.86 -0.60 2 4 86.71 86.71 4 3
4 25 0.02 -0.02 4 6 132.96 132.96 4 3
5 19 1.97 -1.38 2 4 54.38 54.38 6 2
6 19 0.45 -0.32 3 4 84.58 84.58 8 1
7 21 1.20 -0.84 2 4 52.93 52.93 0 5
8 14 -1.03 0.72 0 6 61.82 61.82 0 2
9 20 3.15 -2.21 0 2 32.67 32.67 1 3
10 21 1.59 -1.11 3 6 104.06 104.06 1 3
11 28 6.00 -4.20 0 2 12.47 12.47 8 3
12 9 -1.03 0.72 4 2 88.99 88.99 0 0
13 22 4.44 -3.10 1 2 21.26 21.26 6 2
14 20 5.18 -3.63 1 1 12.03 12.03 2 3
15 13 1.20 -0.84 3 3 77.76 77.76 2 1
16 6 1.69 -1.18 0 0 0.00 0.00 0 1
17 7 2.00 -1.40 0 0 0.00 0.00 0 1
18 3 -0.00 0.00 1 1 20.23 20.23 0 0
19 12 -3.22 2.25 5 6 110.38 110.38 1 1
20 5 -0.97 0.68 2 2 63.32 63.32 1 0
21 18 5.55 -3.89 1 1 37.30 37.30 14 0
AromaticRings Aliphatic_Rings Formal_Charge BertzCT FractionCsp3 \
0 1 0 0 343.22 0.5
1 1 0 0 324.89 0.5
2 1 0 0 253.30 0.5
3 1 2 0 661.30 0.5
4 1 2 0 735.79 0.5
5 2 0 0 523.04 0.5
6 1 0 0 390.18 0.5
7 1 4 0 670.24 0.5
8 2 0 0 616.53 0.5
9 2 1 0 694.88 0.5
10 2 1 0 806.30 0.5
11 3 0 0 880.74 0.5
12 0 0 0 126.92 0.5
13 2 0 0 566.71 0.5
14 2 1 0 624.14 0.5
15 1 0 0 354.63 0.5
16 1 0 0 71.96 0.5
17 1 0 0 129.97 0.5
18 0 0 0 2.75 0.5
19 0 1 0 146.22 0.5
20 0 0 0 42.91 0.5
21 0 0 0 177.94 0.5
NumHeteroatoms MolMR
0 4 44.71
1 2 61.03
2 3 42.41
3 7 85.80
4 9 90.69
5 4 76.38
6 5 73.98
7 4 77.58
8 6 51.20
9 4 81.81
10 6 71.30
11 2 119.58
12 5 36.46
13 5 79.80
14 3 85.78
15 4 46.44
16 0 26.44
17 0 31.18
18 1 12.76
19 6 35.99
20 3 16.69
21 2 77.95
[17:16:52] SMILES Parse Error: syntax error while parsing: GIVEQCCTSICSLYQLENYCN
[17:16:52] SMILES Parse Error: check for mistakes around position 1:
[17:16:52] GIVEQCCTSICSLYQLENYCN
[17:16:52] ^
[17:16:52] SMILES Parse Error: Failed parsing SMILES 'GIVEQCCTSICSLYQLENYCN' for input: 'GIVEQCCTSICSLYQLENYCN'
2. Lipinski’s Rule of Five とその拡張
Lipinski’s Rule of Fiveは経口薬物の薬らしさを評価する最も基本的なルールです。また、近年提案されている拡張ルールについても学習します。
|
|
Lipinski's Rule of Five と拡張ルールによる評価:
======================================================================
薬物分子の評価:
--------------------------------------------------
アスピリン:
Lipinskiスコア: 4/4
拡張スコア: 7/7
Lead-likeスコア: 4/4
分子量: 180.2 (✓)
LogP: 1.31 (✓)
TPSA: 63.6 (✓)
イブプロフェン:
Lipinskiスコア: 4/4
拡張スコア: 7/7
Lead-likeスコア: 3/4
分子量: 206.3 (✓)
LogP: 3.07 (✓)
TPSA: 37.3 (✓)
パラセタモール:
Lipinskiスコア: 4/4
拡張スコア: 7/7
Lead-likeスコア: 4/4
分子量: 151.2 (✓)
LogP: 1.35 (✓)
TPSA: 49.3 (✓)
ペニシリンG:
Lipinskiスコア: 4/4
拡張スコア: 7/7
Lead-likeスコア: 4/4
分子量: 334.4 (✓)
LogP: 0.86 (✓)
TPSA: 86.7 (✓)
アモキシシリン:
Lipinskiスコア: 4/4
拡張スコア: 7/7
Lead-likeスコア: 3/4
分子量: 365.4 (✓)
LogP: 0.02 (✓)
TPSA: 133.0 (✓)
プロプラノロール:
Lipinskiスコア: 4/4
拡張スコア: 7/7
Lead-likeスコア: 4/4
分子量: 260.3 (✓)
LogP: 1.97 (✓)
TPSA: 54.4 (✓)
アテノロール:
Lipinskiスコア: 4/4
拡張スコア: 7/7
Lead-likeスコア: 4/4
分子量: 266.3 (✓)
LogP: 0.45 (✓)
TPSA: 84.6 (✓)
モルヒネ:
Lipinskiスコア: 4/4
拡張スコア: 7/7
Lead-likeスコア: 4/4
分子量: 285.3 (✓)
LogP: 1.20 (✓)
TPSA: 52.9 (✓)
カフェイン:
Lipinskiスコア: 4/4
拡張スコア: 7/7
Lead-likeスコア: 3/4
分子量: 194.2 (✓)
LogP: -1.03 (✓)
TPSA: 61.8 (✓)
ジアゼパム:
Lipinskiスコア: 4/4
拡張スコア: 7/7
Lead-likeスコア: 3/4
分子量: 284.7 (✓)
LogP: 3.15 (✓)
TPSA: 32.7 (✓)
ドキソルビシン:
Lipinskiスコア: 4/4
拡張スコア: 7/7
Lead-likeスコア: 4/4
分子量: 286.2 (✓)
LogP: 1.59 (✓)
TPSA: 104.1 (✓)
タモキシフェン:
Lipinskiスコア: 3/4
拡張スコア: 6/7
Lead-likeスコア: 2/4
分子量: 371.5 (✓)
LogP: 6.00 (✗)
TPSA: 12.5 (✓)
メトホルミン:
Lipinskiスコア: 4/4
拡張スコア: 7/7
Lead-likeスコア: 2/4
分子量: 129.2 (✓)
LogP: -1.03 (✓)
TPSA: 89.0 (✓)
フルオキセチン:
Lipinskiスコア: 4/4
拡張スコア: 7/7
Lead-likeスコア: 3/4
分子量: 309.3 (✓)
LogP: 4.44 (✓)
TPSA: 21.3 (✓)
セルトラリン:
Lipinskiスコア: 3/4
拡張スコア: 6/7
Lead-likeスコア: 3/4
分子量: 306.2 (✓)
LogP: 5.18 (✗)
TPSA: 12.0 (✓)
非薬物分子の評価:
--------------------------------------------------
カフェイン酸: Lipinskiスコア 4/4
ベンゼン: Lipinskiスコア 4/4
トルエン: Lipinskiスコア 4/4
エタノール: Lipinskiスコア 4/4
グルコース: Lipinskiスコア 4/4
アミノ酸_グリシン: Lipinskiスコア 4/4
脂肪酸_パルミチン酸: Lipinskiスコア 3/4
3. QED (Quantitative Estimate of Drug-likeness)
QEDは複数の分子記述子を統合して薬らしさを0-1の値で定量化する手法です。Lipinskiルールよりも洗練された評価が可能です。
|
|
QED (Quantitative Estimate of Drug-likeness) 解析:
============================================================
QEDスコア順ランキング:
----------------------------------------
0.852 - フルオキセチン (薬物)
0.834 - プロプラノロール (薬物)
0.822 - イブプロフェン (薬物)
0.806 - セルトラリン (薬物)
0.798 - ペニシリンG (薬物)
0.792 - ジアゼパム (薬物)
0.703 - モルヒネ (薬物)
0.638 - アテノロール (薬物)
0.595 - パラセタモール (薬物)
0.587 - ドキソルビシン (薬物)
0.553 - アモキシシリン (薬物)
0.550 - アスピリン (薬物)
0.538 - カフェイン (薬物)
0.472 - カフェイン酸 (非薬物)
0.459 - トルエン (非薬物)
0.451 - タモキシフェン (薬物)
0.443 - ベンゼン (非薬物)
0.421 - アミノ酸_グリシン (非薬物)
0.413 - 脂肪酸_パルミチン酸 (非薬物)
0.407 - エタノール (非薬物)
0.290 - グルコース (非薬物)
0.249 - メトホルミン (薬物)
[17:16:53] SMILES Parse Error: syntax error while parsing: GIVEQCCTSICSLYQLENYCN
[17:16:53] SMILES Parse Error: check for mistakes around position 1:
[17:16:53] GIVEQCCTSICSLYQLENYCN
[17:16:53] ^
[17:16:53] SMILES Parse Error: Failed parsing SMILES 'GIVEQCCTSICSLYQLENYCN' for input: 'GIVEQCCTSICSLYQLENYCN'
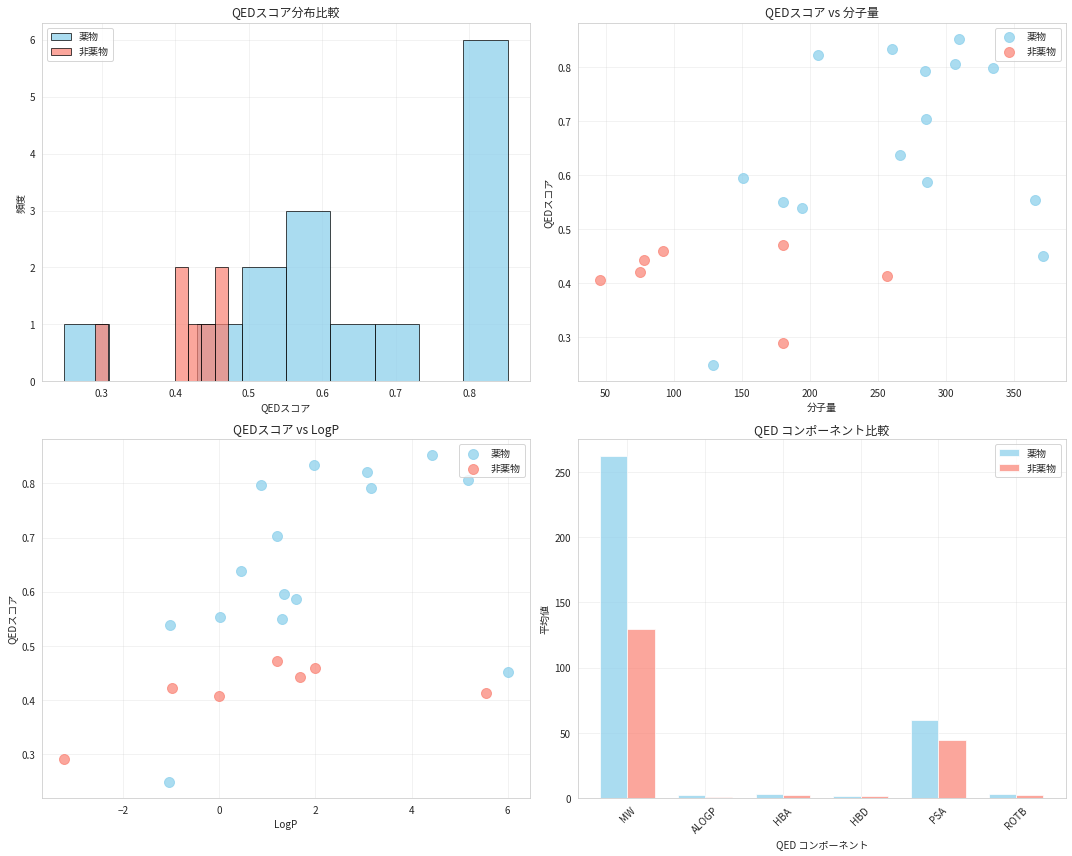
統計情報:
薬物のQEDスコア: 0.651 ± 0.172
非薬物のQEDスコア: 0.415 ± 0.060
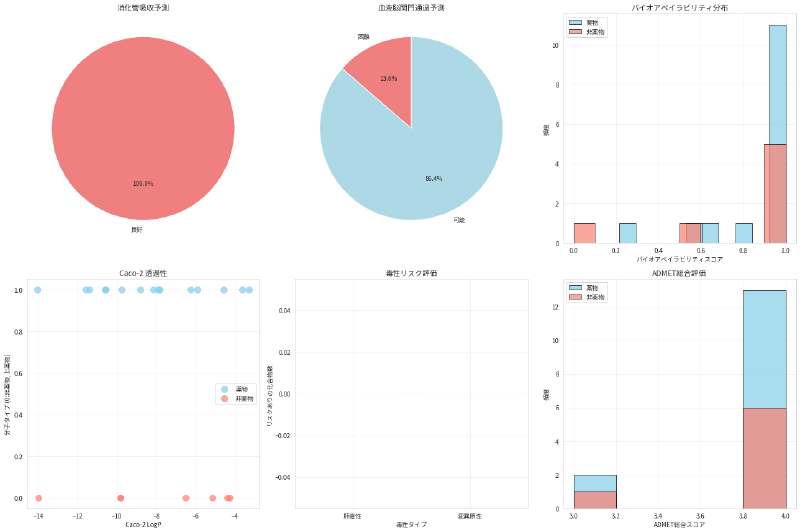
4. ADMET パラメータの予測
ADMET(Absorption, Distribution, Metabolism, Excretion, Toxicity)は薬物の体内動態と毒性を表すパラメータです。分子記述子から簡易的なADMET予測を行います。
|
|
ADMET (Absorption, Distribution, Metabolism, Excretion, Toxicity) 解析:
================================================================================
薬物分子のADMET評価:
------------------------------------------------------------
アスピリン:
消化管吸収予測: 良好
血液脳関門通過: 可能
肝毒性リスク: なし
バイオアベイラビリティ: 1.00
イブプロフェン:
消化管吸収予測: 良好
血液脳関門通過: 可能
肝毒性リスク: なし
バイオアベイラビリティ: 1.00
パラセタモール:
消化管吸収予測: 良好
血液脳関門通過: 可能
肝毒性リスク: なし
バイオアベイラビリティ: 1.00
ペニシリンG:
消化管吸収予測: 良好
血液脳関門通過: 可能
肝毒性リスク: なし
バイオアベイラビリティ: 1.00
アモキシシリン:
消化管吸収予測: 良好
血液脳関門通過: 困難
肝毒性リスク: なし
バイオアベイラビリティ: 0.68
プロプラノロール:
消化管吸収予測: 良好
血液脳関門通過: 可能
肝毒性リスク: なし
バイオアベイラビリティ: 1.00
アテノロール:
消化管吸収予測: 良好
血液脳関門通過: 可能
肝毒性リスク: なし
バイオアベイラビリティ: 1.00
モルヒネ:
消化管吸収予測: 良好
血液脳関門通過: 可能
肝毒性リスク: なし
バイオアベイラビリティ: 1.00
カフェイン:
消化管吸収予測: 良好
血液脳関門通過: 可能
肝毒性リスク: なし
バイオアベイラビリティ: 1.00
ジアゼパム:
消化管吸収予測: 良好
血液脳関門通過: 可能
肝毒性リスク: なし
バイオアベイラビリティ: 1.00
ドキソルビシン:
消化管吸収予測: 良好
血液脳関門通過: 困難
肝毒性リスク: なし
バイオアベイラビリティ: 1.00
タモキシフェン:
消化管吸収予測: 良好
血液脳関門通過: 可能
肝毒性リスク: なし
バイオアベイラビリティ: 0.21
メトホルミン:
消化管吸収予測: 良好
血液脳関門通過: 可能
肝毒性リスク: なし
バイオアベイラビリティ: 1.00
フルオキセチン:
消化管吸収予測: 良好
血液脳関門通過: 可能
肝毒性リスク: なし
バイオアベイラビリティ: 0.82
セルトラリン:
消化管吸収予測: 良好
血液脳関門通過: 可能
肝毒性リスク: なし
バイオアベイラビリティ: 0.59
[17:16:53] SMILES Parse Error: syntax error while parsing: GIVEQCCTSICSLYQLENYCN
[17:16:53] SMILES Parse Error: check for mistakes around position 1:
[17:16:53] GIVEQCCTSICSLYQLENYCN
[17:16:53] ^
[17:16:53] SMILES Parse Error: Failed parsing SMILES 'GIVEQCCTSICSLYQLENYCN' for input: 'GIVEQCCTSICSLYQLENYCN'
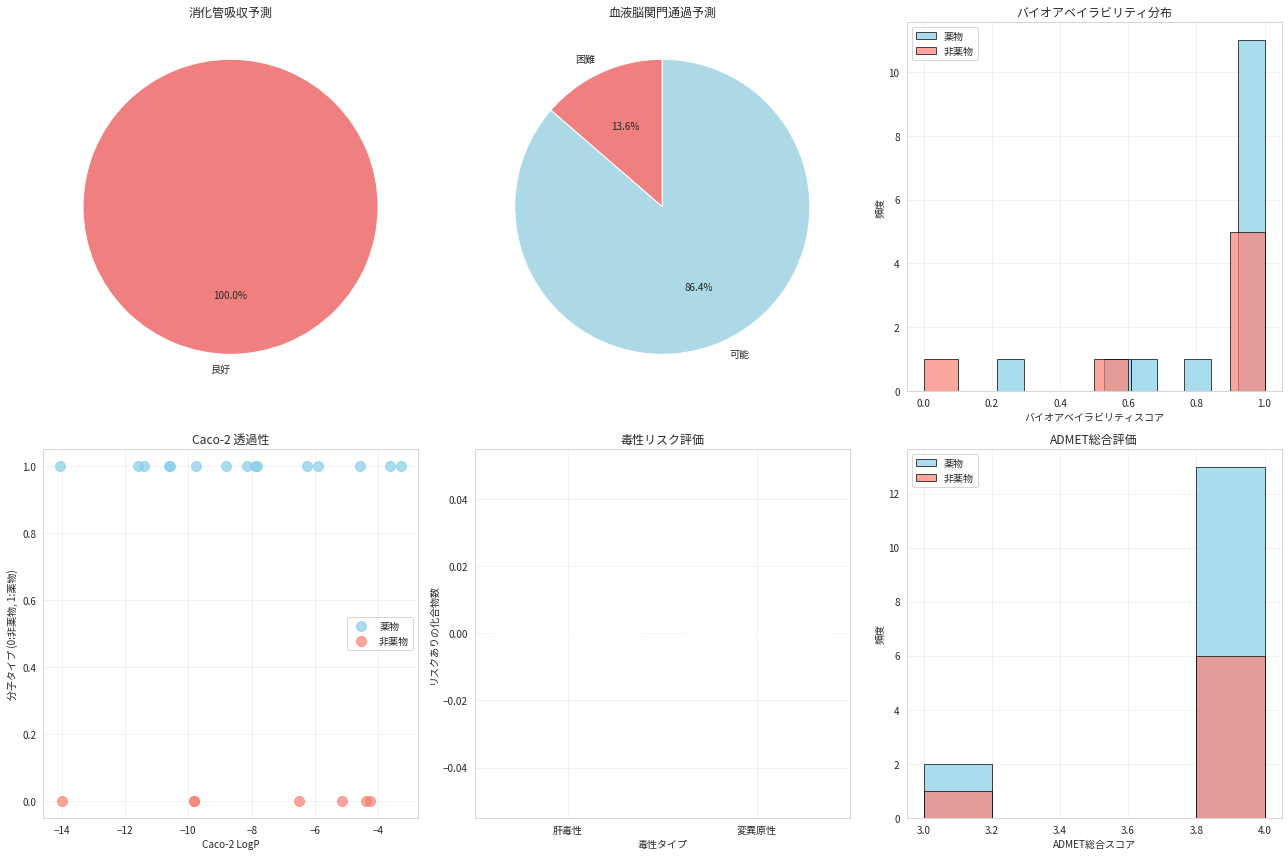
ADMET統計:
薬物のADMET平均スコア: 3.87 ± 0.35
非薬物のADMET平均スコア: 3.86 ± 0.38
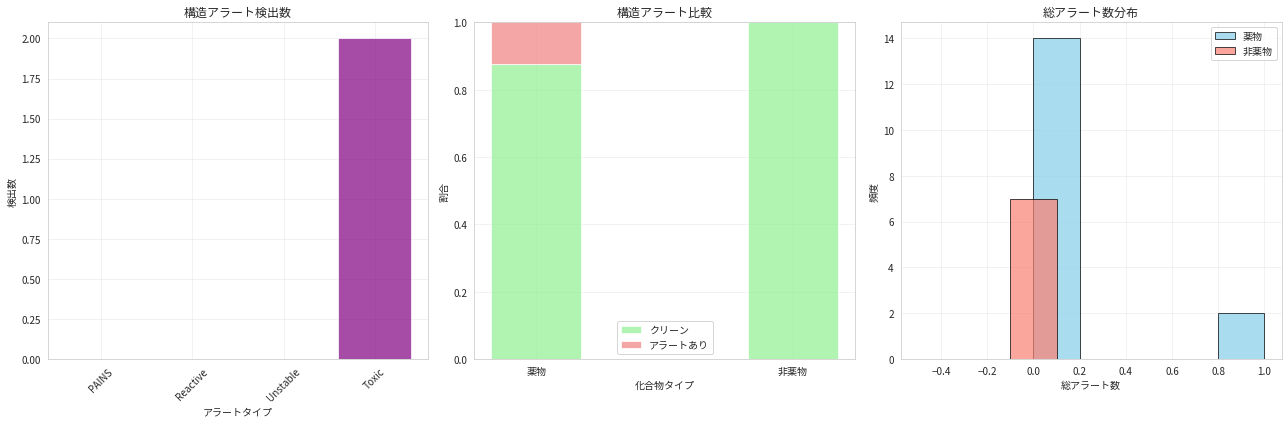
5. 構造アラートとフィルタリング
特定の部分構造は毒性や副作用と関連があることが知られています。これらの「構造アラート」を検出する手法を学習します。
|
|
構造アラート解析:
==================================================
構造アラートを持つ化合物:
----------------------------------------
ペニシリンG (薬物):
PAINS: 0, 反応性: 0, 不安定: 0, 毒性: 1
アモキシシリン (薬物):
PAINS: 0, 反応性: 0, 不安定: 0, 毒性: 1
クリーンな化合物数: 21/23
[17:16:54] SMILES Parse Error: syntax error while parsing: GIVEQCCTSICSLYQLENYCN
[17:16:54] SMILES Parse Error: check for mistakes around position 1:
[17:16:54] GIVEQCCTSICSLYQLENYCN
[17:16:54] ^
[17:16:54] SMILES Parse Error: Failed parsing SMILES 'GIVEQCCTSICSLYQLENYCN' for input: 'GIVEQCCTSICSLYQLENYCN'
6. 機械学習による薬物・非薬物分類
計算した記述子を用いて、機械学習モデルで薬物と非薬物を分類する手法を学習します。
|
|
機械学習による薬物・非薬物分類:
==================================================
特徴量数: 17
サンプル数: 22
薬物: 15個, 非薬物: 7個
分類結果:
------------------------------
Classification Report:
precision recall f1-score support
非薬物 0.33 0.50 0.40 2
薬物 0.75 0.60 0.67 5
accuracy 0.57 7
macro avg 0.54 0.55 0.53 7
weighted avg 0.63 0.57 0.59 7
混同行列:
[[1 1]
[2 3]]
特徴量重要度 (上位10位):
Feature Importance
0 分子量 0.192701
1 Heavy_Atoms 0.181343
13 MolMR 0.165559
14 QED_Score 0.153368
10 BertzCT 0.096474
5 TPSA 0.042939
2 LogP 0.041389
7 RingCount 0.040413
3 HBD 0.023538
8 AromaticRings 0.021270
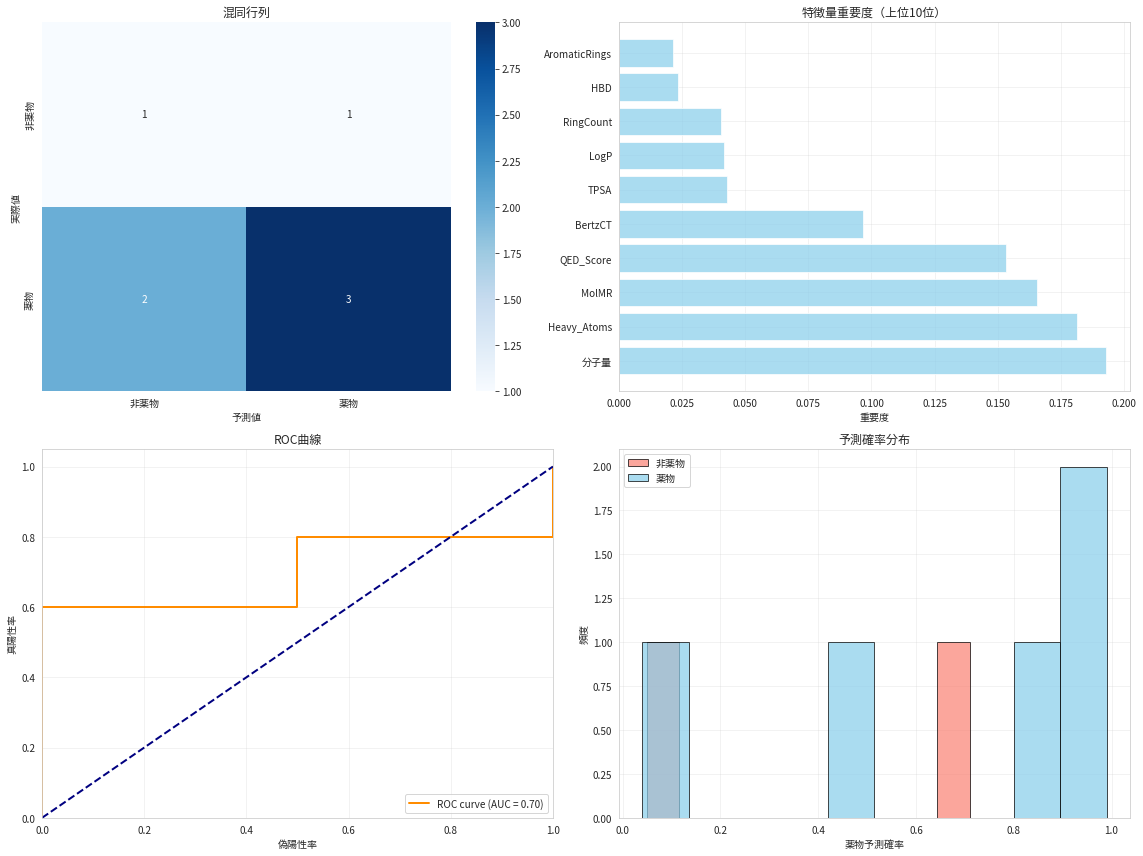
モデル性能:
AUC: 0.700
精度: 0.571
7. 薬らしさスコアの統合評価
これまで計算した各種指標を統合して、包括的な薬らしさスコアを作成します。
|
|
統合薬らしさスコア:
============================================================
統合薬らしさスコア ランキング:
--------------------------------------------------
1. 0.956 - フルオキセチン (薬物)
2. 0.950 - プロプラノロール (薬物)
3. 0.946 - イブプロフェン (薬物)
4. 0.937 - ジアゼパム (薬物)
5. 0.911 - モルヒネ (薬物)
6. 0.899 - ペニシリンG (薬物)
7. 0.891 - アテノロール (薬物)
8. 0.879 - セルトラリン (薬物)
9. 0.879 - パラセタモール (薬物)
10. 0.865 - アスピリン (薬物)
11. 0.862 - カフェイン (薬物)
12. 0.841 - カフェイン酸 (非薬物)
13. 0.838 - トルエン (非薬物)
14. 0.833 - ベンゼン (非薬物)
15. 0.826 - アミノ酸_グリシン (非薬物)
16. 0.822 - エタノール (非薬物)
17. 0.814 - ドキソルビシン (薬物)
18. 0.775 - メトホルミン (薬物)
19. 0.773 - タモキシフェン (薬物)
20. 0.763 - アモキシシリン (薬物)
21. 0.762 - 脂肪酸_パルミチン酸 (非薬物)
22. 0.725 - グルコース (非薬物)
スコア成分詳細(上位5位):
------------------------------------------------------------
フルオキセチン:
統合スコア: 0.956
QED: 0.852
ADMET: 1.000
Lipinski: 1.000
Alerts: 1.000
プロプラノロール:
統合スコア: 0.950
QED: 0.834
ADMET: 1.000
Lipinski: 1.000
Alerts: 1.000
イブプロフェン:
統合スコア: 0.946
QED: 0.822
ADMET: 1.000
Lipinski: 1.000
Alerts: 1.000
ジアゼパム:
統合スコア: 0.937
QED: 0.792
ADMET: 1.000
Lipinski: 1.000
Alerts: 1.000
モルヒネ:
統合スコア: 0.911
QED: 0.703
ADMET: 1.000
Lipinski: 1.000
Alerts: 1.000
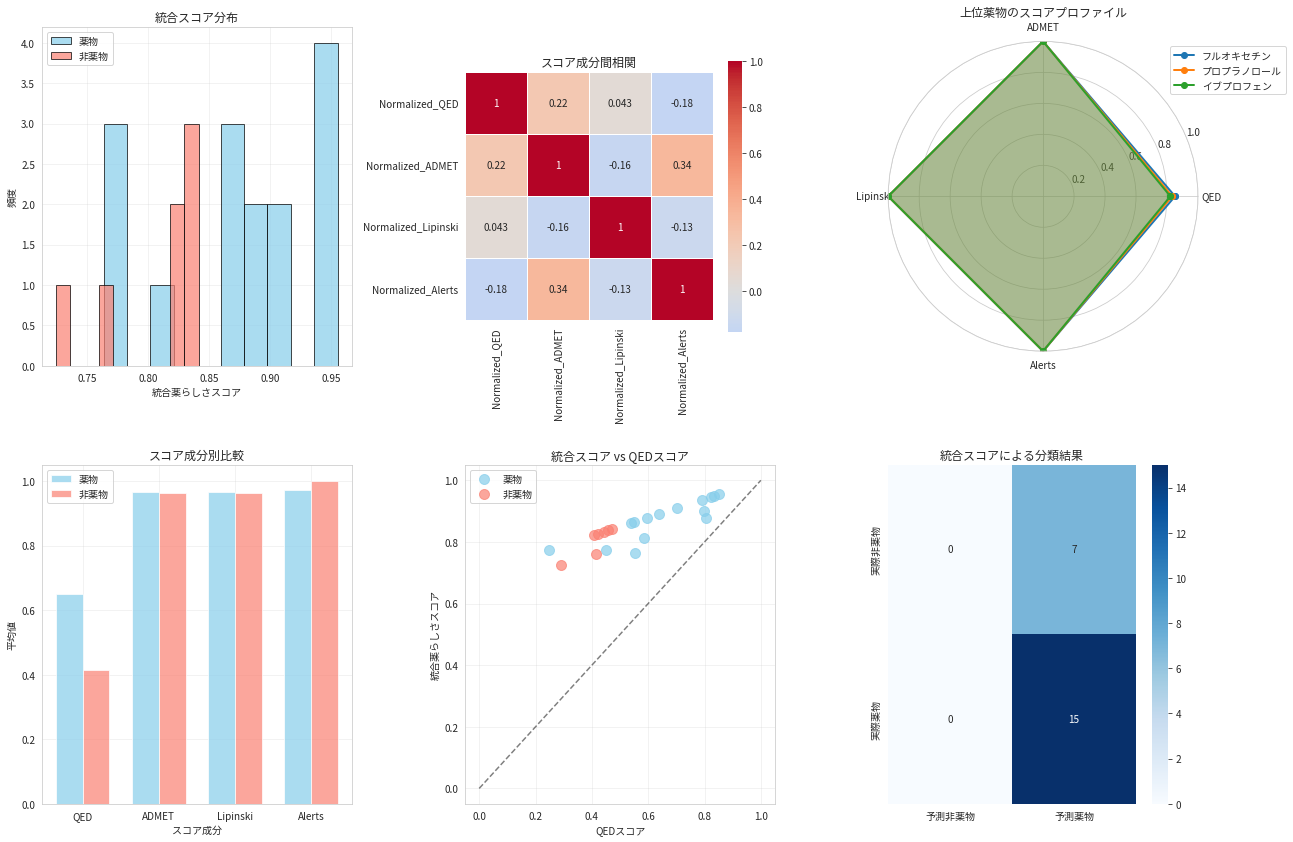
統合スコア分類性能 (閾値: 0.5):
精度: 0.682
適合率: 0.682
再現率: 1.000
F1スコア: 0.811
統計情報:
薬物の統合スコア: 0.873 ± 0.066
非薬物の統合スコア: 0.807 ± 0.045
まとめ
本ノートブックでは、記述子による薬らしさの表現について包括的に学習しました:
主要なポイント
-
基本的な薬物記述子:
- 分子量、LogP、TPSA、水素結合数など
- 物理化学的性質の数値化
-
Lipinski’s Rule of Five:
- 経口薬物の基本的な判定基準
- 拡張ルールとLead-like基準
-
QED (Quantitative Estimate of Drug-likeness):
- 複数記述子を統合した薬らしさ評価
- 0-1スケールでの定量化
-
ADMET パラメータ:
- 吸収・分布・代謝・排泄・毒性の予測
- 薬物動態の初期評価
-
構造アラート:
- PAINS、反応性、毒性パターンの検出
- 好ましくない部分構造の特定
-
機械学習による分類:
- 薬物・非薬物の自動判別
- 特徴量重要度の分析
-
統合薬らしさスコア:
- 複数指標の重み付け統合
- 包括的な薬らしさ評価
創薬への応用
- 初期スクリーニング: 薬らしさフィルタによる候補化合物の絞り込み
- リード最適化: 記述子を指標とした構造修飾
- バーチャルライブラリ設計: 薬らしさを考慮した合成計画
- 開発可能性評価: ADMET予測による開発リスク評価
次のステップ
- より高精度なADMET予測モデルの構築
- 疾患特異的な薬らしさ基準の開発
- 3D構造を考慮した薬らしさ評価
- AI創薬における薬らしさ制約の組み込み
|
|
記述子による薬らしさ表現学習の統計情報:
============================================================
解析化合物数: 22
薬物: 15個
非薬物: 7個
計算記述子数: 17
各指標の平均値:
----------------------------------------
QEDスコア: 0.576
ADMETスコア: 0.966
Lipinskiスコア: 0.966
構造アラート(逆数): 0.982
統合薬らしさスコア: 0.852
手法の特徴:
------------------------------
• Lipinski's Rule: 経口薬物の基本判定基準
• QED: 複数記述子の統合評価
• ADMET: 薬物動態・毒性予測
• 構造アラート: 望ましくない部分構造の検出
• 機械学習: データ駆動型の薬らしさ判定
• 統合スコア: 包括的な薬らしさ定量化
学習が完了しました!